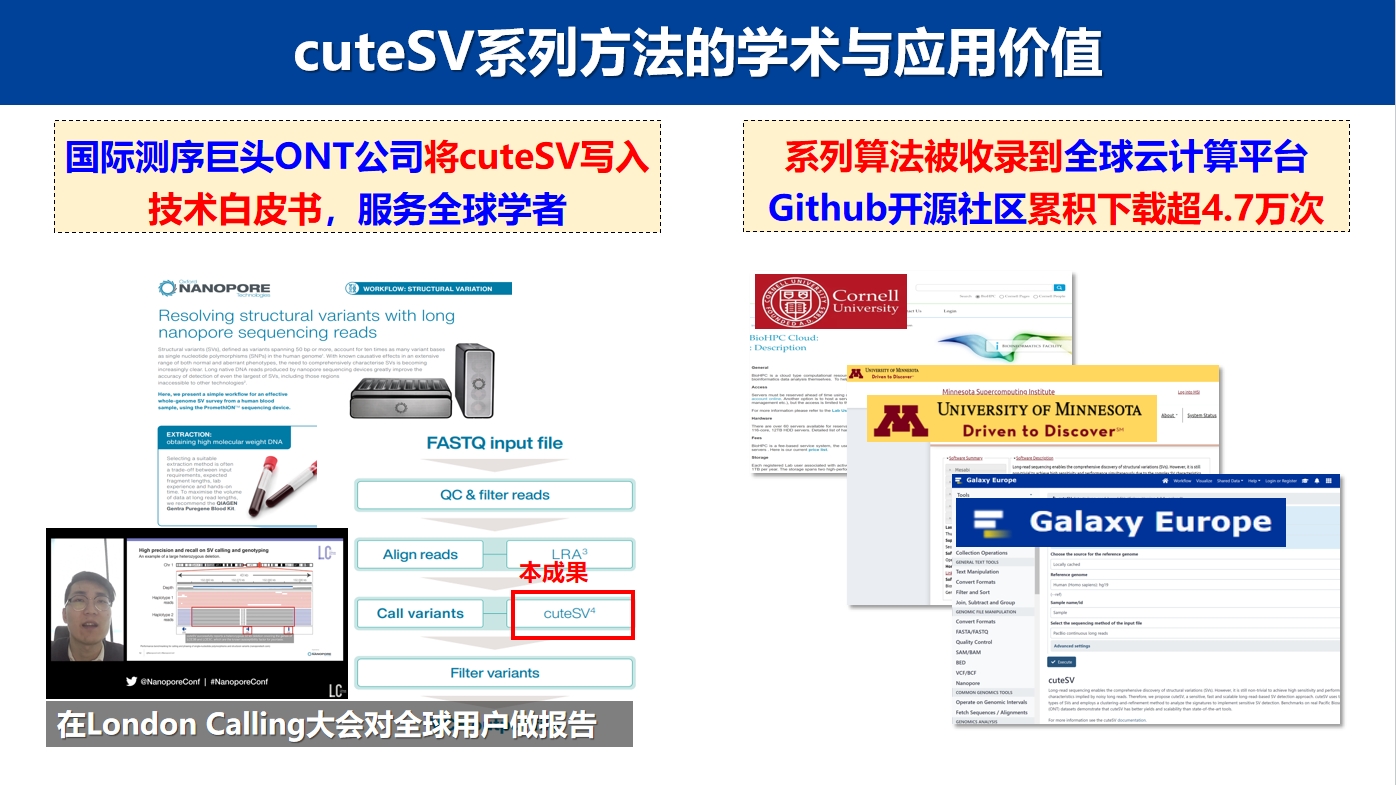
基因组序列非线性表示理论
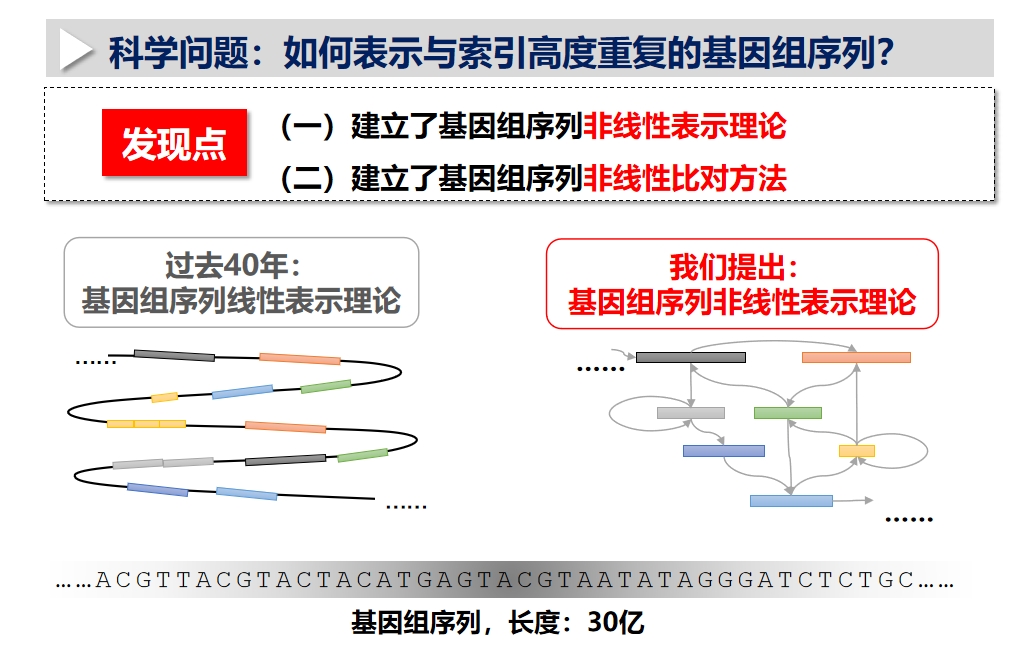
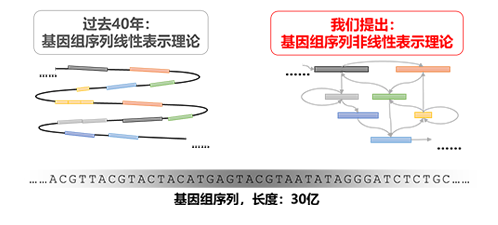
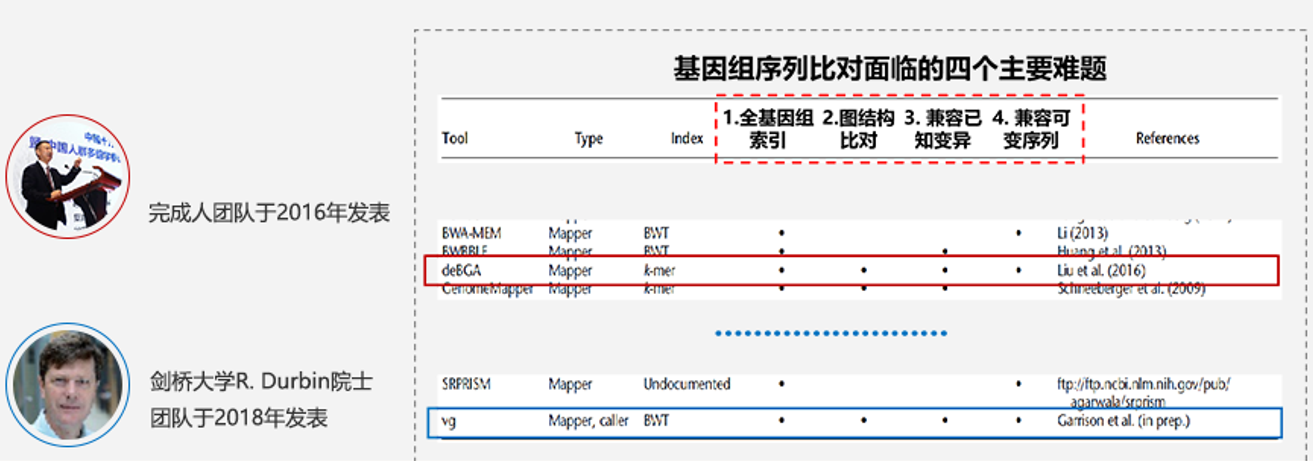
(一)建立了基因组序列非线性表示理论
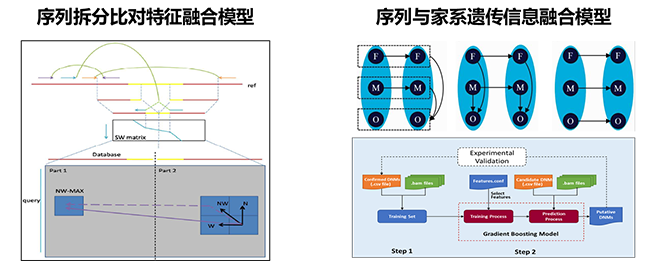
(二)建立了基因组序列非线性比对方法
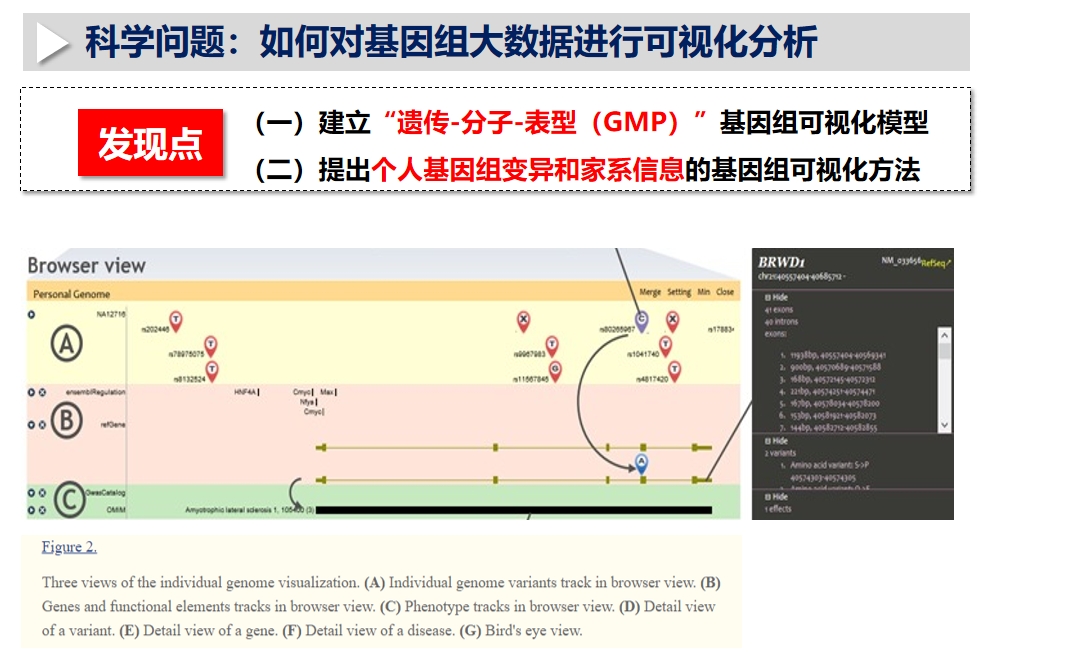
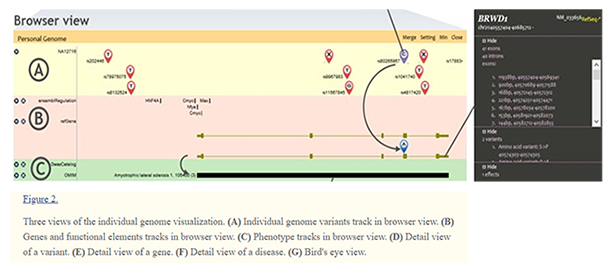
(三)实现了对个人和家系基因组大数据内在特性的系统展示和对个人和家系基因组大数据的交互式分析
(一)建立了基因组序列非线性表示理论
(二)建立了基因组序列非线性比对方法
(三)实现了对个人和家系基因组大数据内在特性的系统展示和对个人和家系基因组大数据的交互式分析